-
Notifications
You must be signed in to change notification settings - Fork 1
Examples_ru
Область применения разработанной библиотеки извлечения признаков из метагеномных данных не ограничивается анализом образцов микробиоты кишечника человека. Ниже представлены два примера: анализ водных образцов и анализ микробиоты буйволов.
Для примера из открытых источников были отобраны метагеномные образцы воды, взятые из четырех разных водоемов. При этом для секвенирования из образцов был отобран виром – множество всех вирусных частиц, присутствующих в образцах.
Для анализа было использовано 28 образцов, которые могут быть загружены из базы данных NCBI по следующим идентификаторам: [SRR013515, SRR013516, SRR013521, DRR147672, DRR147675, DRR147676, DRR147677, DRR147678, DRR147679, DRR147680, DRR147681, DRR147682, SRR2147000, SRR4447083, SRR4449190, SRR4449206, SRR4449214, SRR4449225, SRR4449240, SRR4449244, SRR4449250, SRR4449256, SRR4449268, SRR4449292, SRR1296481, SRR1301999, SRR1302010, SRR1302020]. Предполагается, что файлы загружены в папку data/. Эти же данные продублированы для загрузки по адресу https://ctlab.itmo.ru/~aivanov/example_data/water/.
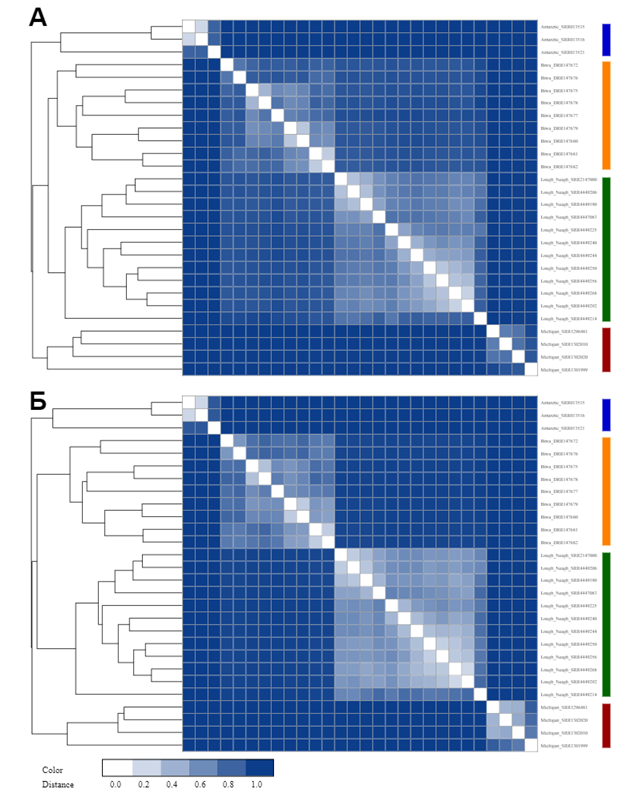
Данные были проанализированы с помощью двух модулей библиотеки MetaFX: алгоритма metafast для извлечения признаков и кластеризации без учителя, а также алгоритма unique для извлечения признаков с использованием места взятия образцов в качестве классов. На основе полученных матриц признаков в программе MetaFast была построена иерархическая кластеризация всех образцов с использованием несходства Брея-Кертиса для оценки похожести двух образцов. Результаты представлены на рисунке ниже. Цветами справа от рисунка обозначены группы образцов (синий – Лимнополар, оранжевый – Бива, зеленый – Лох-Ней, красный – Мичиган). Оба метода хорошо справились с кластеризацией, корректно сгруппировав образцы по месту взятия проб. При этом заметно, что метод на основе уникальных k‑меров уменьшил расстояния внутри кластеров и увеличил расстояния между кластерами за счет использования информации о метках образцов на этапе извлечения признаков.
Для воспроизведения результатов могут быть использованы следующие команды:
metafx metafast -t 64 -m 400G -w wd_metafast -k 31 -i data/*.fastq.gz
metafx unique -t 32 -m 200G -w wd_unique -k 31 -i sample_list.txt --kmers-dir wd_metafast/kmer-counter-many/kmers/
mkdir vectors_unique
while read line; do cat wd_unique/features_Antarctic/vectors/$line.vec wd_unique/features_Biwa/vectors/$line.vec wd_unique/features_Lough_Neagh/vectors/$line.vec wd_unique/features_Michigan/vectors/$line.vec > vectors_unique/$line.vec; done<<<$(cut -f1 wd_unique/samples_categories.tsv)
metafast.sh -t dist-matrix-calculator -p 12 -m 8G -w wd_matrix_unique --features vectors_unique/*.vec
metafast.sh -t heatmap-maker -p 12 -m 8G -w wd_matrix_unique -i wd_matrix_unique/dist_matrix*.txt
Содержимое файла sample_list.txt:
data/SRR013515.fastq.gz Antarctic
data/SRR013516.fastq.gz Antarctic
data/SRR013521.fastq.gz Antarctic
data/DRR147672.fastq.gz Biwa
data/DRR147675.fastq.gz Biwa
data/DRR147676.fastq.gz Biwa
data/DRR147677.fastq.gz Biwa
data/DRR147678.fastq.gz Biwa
data/DRR147679.fastq.gz Biwa
data/DRR147680.fastq.gz Biwa
data/DRR147681.fastq.gz Biwa
data/DRR147682.fastq.gz Biwa
data/SRR2147000.fastq.gz Lough_Neagh
data/SRR4447083.fastq.gz Lough_Neagh
data/SRR4449190.fastq.gz Lough_Neagh
data/SRR4449206.fastq.gz Lough_Neagh
data/SRR4449214.fastq.gz Lough_Neagh
data/SRR4449225.fastq.gz Lough_Neagh
data/SRR4449240.fastq.gz Lough_Neagh
data/SRR4449244.fastq.gz Lough_Neagh
data/SRR4449250.fastq.gz Lough_Neagh
data/SRR4449256.fastq.gz Lough_Neagh
data/SRR4449268.fastq.gz Lough_Neagh
data/SRR4449292.fastq.gz Lough_Neagh
data/SRR1296481.fastq.gz Michigan
data/SRR1301999.fastq.gz Michigan
data/SRR1302010.fastq.gz Michigan
data/SRR1302020.fastq.gz Michigan
Результаты в виде картинок содержатся в файлах wd_metafast/matrices/dist_matrix_*_heatmap.png и wd_matrix_unique/dist_matrix_*_heatmap.png
Помимо анализа образцов вирома воды и микробиоты кишечника человека, актуальным является анализ метагеномных образцов в животноводстве и сельском хозяйстве. В качестве примера был проведен анализ микробиоты желудка самок молочных буйволов.
Для анализа было использовано 15 метагеномных образцов, разбитых на 4 категории по возрасту. Они могут быть загружены из базы данных NCBI по следующим идентификаторам: [SRR14129954, SRR14129955, SRR14129956, SRR14129957, SRR14129958, SRR14129959, SRR14129960, SRR14129961, SRR14129962, SRR14129963, SRR14129964, SRR14129965, SRR14129966, SRR14129967, SRR14129968]. Предполагается, что файлы загружены в папку data/. Эти же данные продублированы для загрузки по адресу https://ctlab.itmo.ru/~aivanov/example_data/buffalo/.
Данные были проанализированы с помощью алгоритма unique библиотеки MetaFast для извлечения признаков с использованием возраста животных в качестве классов. При этом были рассмотрены два сценария работы.
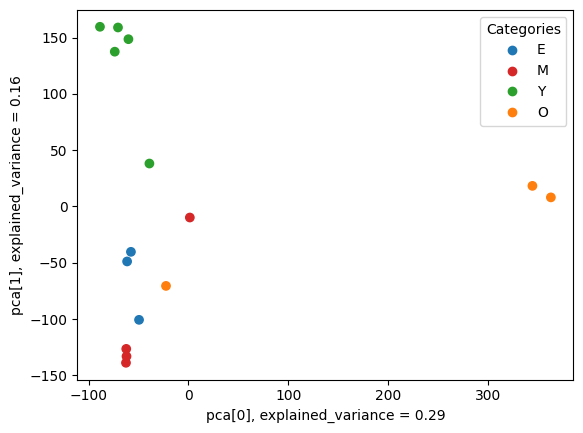
В первом случае анализу подвергались сразу все образцы, и затем была проведена визуализация с использованием метода уменьшения размерности pca. Визуализация представлена на рисунке ниже. Лучше всего кластеризовались образцы молодых особей. В то же время один из образцов самой старшей категории оказался близко к образцам среднего возраста, в то время как остальные расположились ожидаемо далеко.
Для воспроизведения результатов могут быть использованы следующие команды:
metafx extract_kmers -t 32 -m 200G -w wd_kmers_b5 -b 5 -k 31 -i data/*.fastq.gz
metafx unique -t 32 -m 200G -w wd_unique_b5 -k 31 -i labels.tsv --kmers-dir wd_kmers_b5/kmers/ --skip-graph
metafx pca -f wd_unique_b5/feature_table.tsv -i wd_unique_b5/samples_categories.tsv -w wd_pca_b5
Содержимое файла labels.tsv:
data/SRR14129954.fastq.gz E
data/SRR14129955.fastq.gz M
data/SRR14129956.fastq.gz M
data/SRR14129957.fastq.gz M
data/SRR14129958.fastq.gz M
data/SRR14129959.fastq.gz Y
data/SRR14129960.fastq.gz Y
data/SRR14129961.fastq.gz Y
data/SRR14129962.fastq.gz O
data/SRR14129963.fastq.gz O
data/SRR14129964.fastq.gz O
data/SRR14129965.fastq.gz E
data/SRR14129966.fastq.gz E
data/SRR14129967.fastq.gz Y
data/SRR14129968.fastq.gz Y
Во втором случае по одному образцу из каждого класса было отложено в качестве тестовой группы, а признаки извлекались из оставшихся 11 образцов. Затем был применен алгоритм fit_predict из библиотеки MetaFast, который обучил модель случайного леса на извлеченных признаках для 11 образцов и провел классификацию тестовых образцов. Все 11 исходных образцов были верно классифицированы, а из четырех тестовых в двух предсказаниях случились ошибки. При этом необходимо отметить, что степень ошибки не велика, поскольку отклонения произошли в соседнюю возрастную группу, а не перескочили через несколько групп. Один образец получил предсказание Y вместо M, а еще один O вместо E. С учетом очень маленькой выборки данные результаты являются положительными и могут быть улучшены при увеличении числа тренировочных образцов.
Для воспроизведения результатов могут быть использованы следующие команды:
metafx extract_kmers -t 32 -m 200G -w wd_kmers_b5 -b 5 -k 31 -i data/*.fastq.gz
metafx unique -t 32 -m 200G -w wd_unique_b5_train -k 31 -i labels_train.tsv --kmers-dir wd_kmers_b5/kmers/ --skip-graph
metafx fit_predict -f wd_unique_b5_train/feature_table.tsv -i wd_unique_b5_train/samples_categories.tsv -w wd_fp_b5_train
Содержимое файла labels_train.tsv:
data/SRR14129955.fastq.gz M
data/SRR14129956.fastq.gz M
data/SRR14129957.fastq.gz M
data/SRR14129959.fastq.gz Y
data/SRR14129960.fastq.gz Y
data/SRR14129961.fastq.gz Y
data/SRR14129962.fastq.gz O
data/SRR14129963.fastq.gz O
data/SRR14129965.fastq.gz E
data/SRR14129966.fastq.gz E
data/SRR14129967.fastq.gz Y
Полученные результаты для тестовых образцов:
| Образец | Истинный класс | Предсказанный класс |
|---|---|---|
| SRR14129954 | E | O |
| SRR14129958 | M | Y |
| SRR14129964 | O | O |
| SRR14129968 | Y | Y |